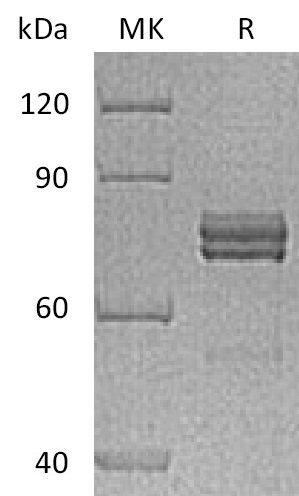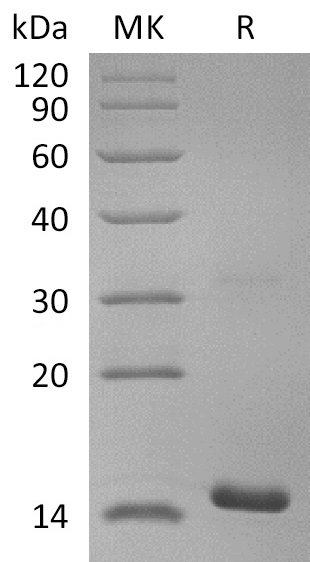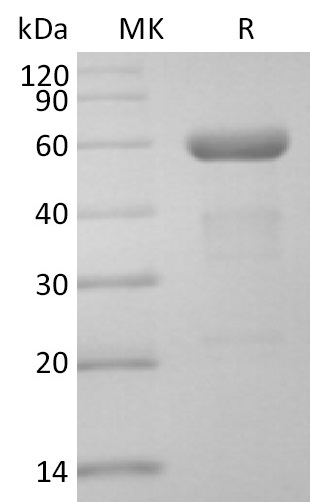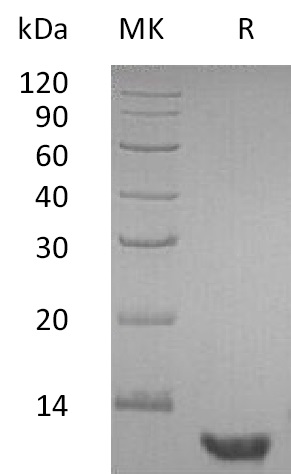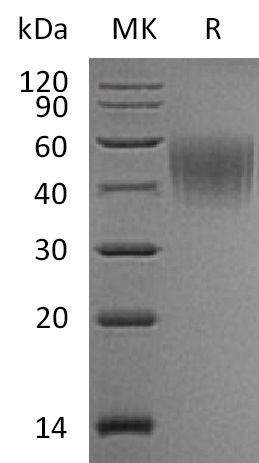

mRNA Cap 2´-O-Methyltransferase, GMP Grade
- GMP-M072-1
- 近岸蛋白
- 上海市
- 现货
- 2500U
- 询价
- 2023-02-03 09:46:49
苏州近岸蛋白质科技股份有限公司
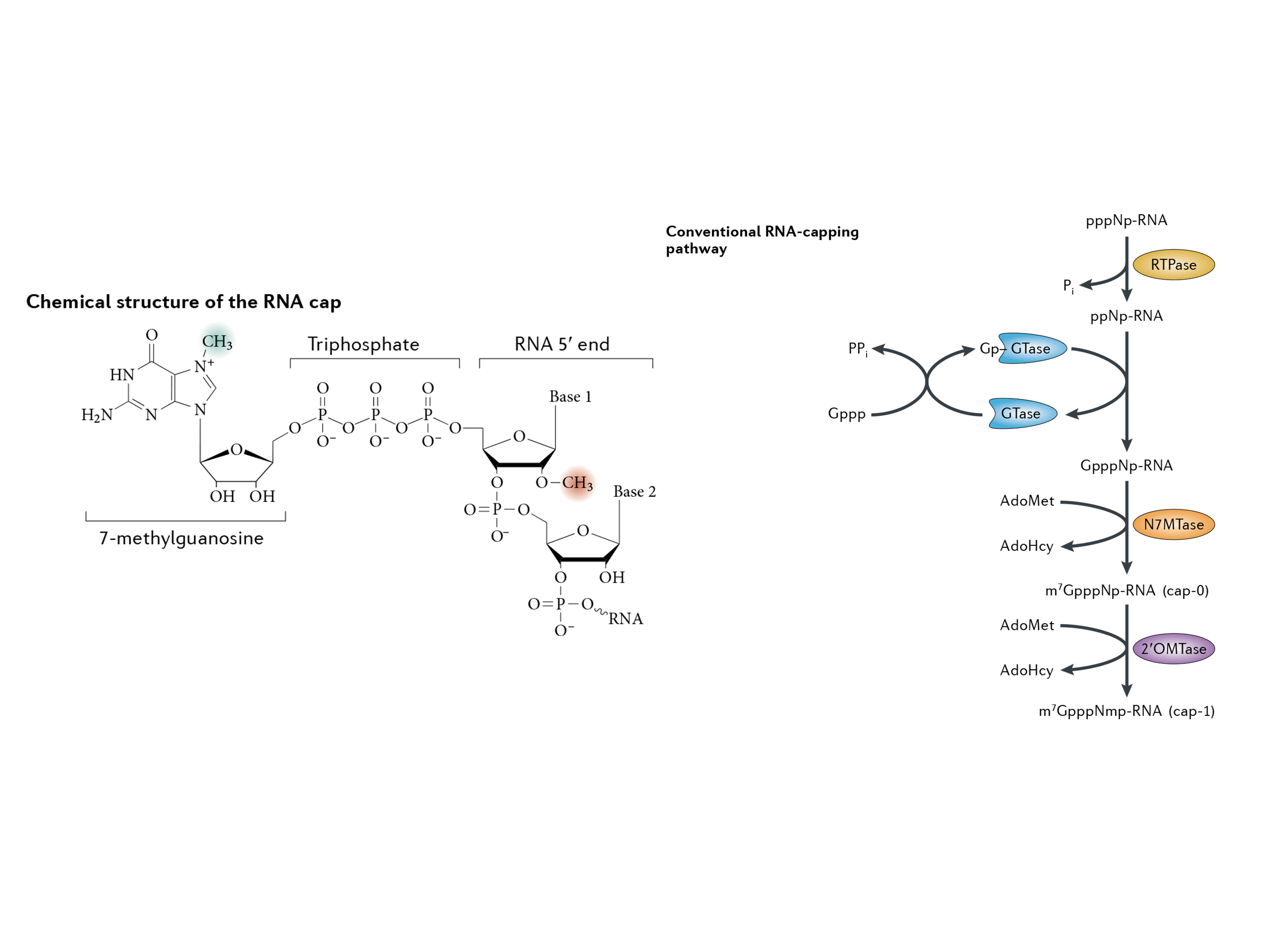
Cap 0结构的mRNA 2´-O甲基化(Cap1),以提高mRNA的翻译和表达效率。
-20±5℃
在37℃下一个小时内将10 pmol 80nt的带帽RNA转录子甲基化所需酶量。
SAM:需事先计算好SAM的用量,在反应开始前把分装的32 mM的储备液稀释成2 mM的工作液。SAM在pH 7-8,37°C条件下不稳定,需要在反应开始之前新鲜配置。为避免SAM降解,该工作液需要保存于冰上。
Cap 0-RNA的制备:IVT RNA需进行纯化并溶解于RNase-free水中,不能将RNA溶解在EDTA溶液或其他盐溶液中。Cap 0-RNA可通过近岸蛋白质Vaccinia Capping System(货号:M062)反应体系获得。为减少反应步骤,也可通过在同一体系内添加两种酶直接获得Cap1-RNA,该反应基于近岸蛋白质Cap 1 Capping System(货号:M082),试剂盒内涵盖Vaccinia Capping System和mRNA Cap 2´-O-Methyltransferase。
RNA二级结构:一些RNA转录本可能形成稳定的二级结构(同源二聚体和发卡结构),如二级结构发生在转录本的5'端会影响加帽效率。在与加帽酶反应之前加热RNA可以去除转录产物5´端的二级结构,如果转录产物的5´端结构复杂,可以把加热时间延长至10min。
Poly(A) 尾:如果加帽的RNA需要添加3'-poly(A)尾巴,可以使用近岸蛋白质E. coli Poly(A) Polymerase(货号:M012)进行加尾。加帽和加尾后的RNA需在进行RNA转染实验前做纯化处理。
RNase Inhibitor:在配置反应体系时,可以加入0.5 µl近岸蛋白质RNase 抑制剂(货号:E125),同时去掉等体积的RNase-free水。
1. 加帽效率低有哪些原因及解决方法?
1) 在加帽反应之前,应将IVT产生的RNA纯化去除残留蛋白质,污染物和未结合核苷酸,并溶解在RNase-free水中,不能将RNA溶解在EDTA或其他盐溶液中;
2) SAM在室温下会慢慢降解,需始终放置在冰上,由于SAM的降解导致的N7甲基化效率低,会进一步导致加帽失败;
3) 可以适当增加RNA热变性的条件,把加热时间延长至10min,加帽反应时间可延长至60min;
4) 某些RNA会在5'末端形成稳定结构(如同源二聚体,发卡结构),限制加帽酶靠近。分析序列后可以将RNA变性温度增加。如5'末端高度结构化,需要通过分子生物学技术来修改序列。通常可以通过在转录RNA(非编码区)的DNA模板前5个碱基中进行单点突变来实现。
2. 缓冲液出现白色沉淀如何解决?
1) 将反应缓冲液在37℃孵育5min,彻底混匀以溶解沉淀物;
2) 不要将试剂盒储存于-70℃。