1区,IF=19.16| 云序RNA乙酰化测序acRIP-seq助力病毒复制机制研究!
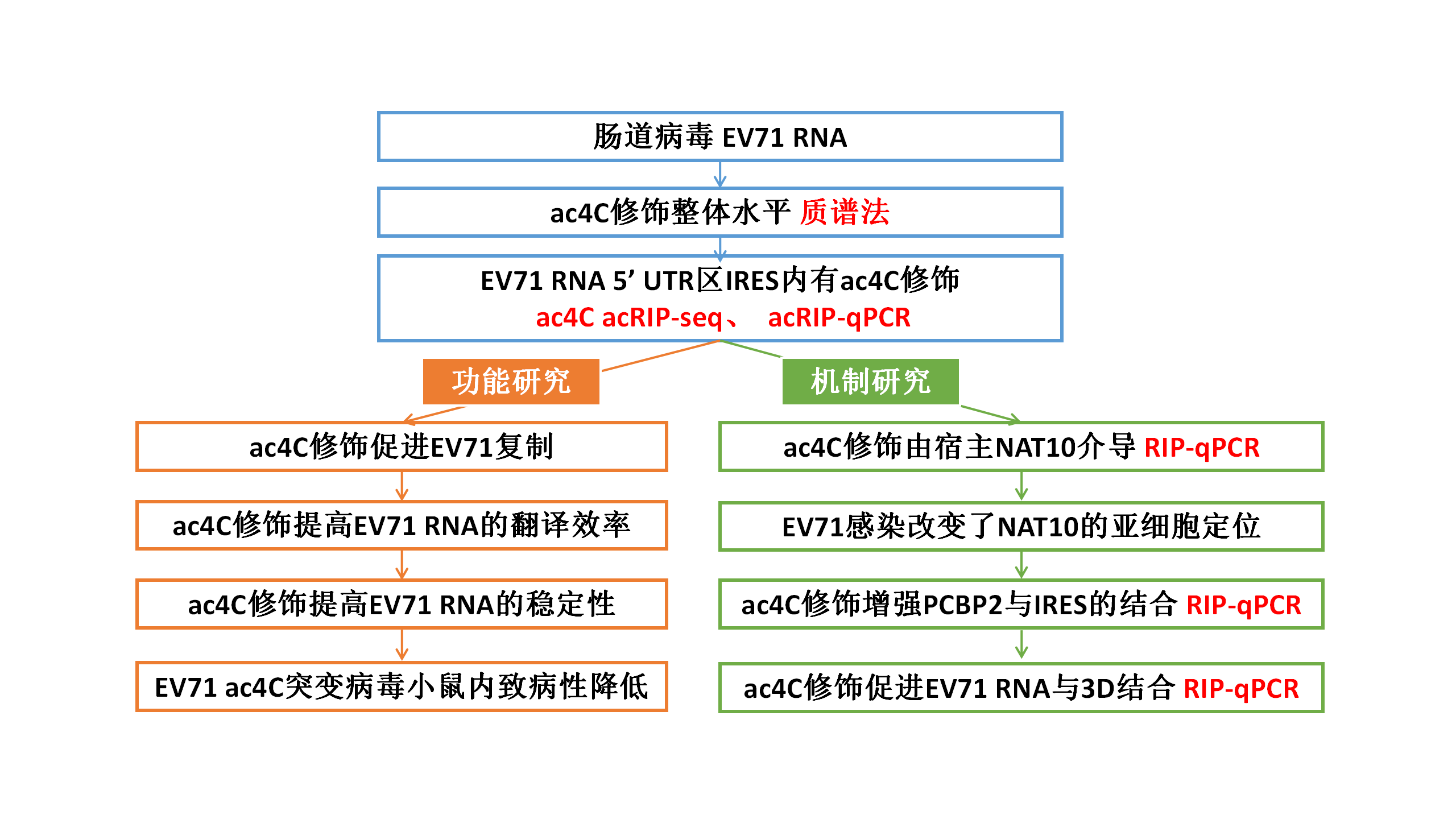
N4-乙酰胞苷(ac4C)对mRNA的翻译和稳定性至关重要。虽然在RNA病毒中发现了ac4C,但ac4C影响病毒复制的详细机制尚不清楚。2022年8月16日,云序客户中国科学院武汉病毒所与深圳大学的研究者们在Nucleic Acids Research(IF=19.16)杂志上发表了文章“N4-acetylcytidine regulates the replication and pathogenicity of enterovirus 71”。本文报道了肠道病毒71(EV71)基因组的5’UTR区存在由宿主乙酰转移酶NAT10催化产生的ac4C修饰。而且,ac4C通过选择性招募PCBP2到IRES来增强病毒RNA的翻译,并提高了RNA的稳定性。此外,抑制NAT10和突变ac4C位点抑制了EV71的复制,且ac4c缺陷突变体EV71在小鼠体内表现出的致病性降低。此项研究结果突出说明了ac4C在EV71感染中的重要作用,并为潜在的抗病毒治疗提供了新的见解。云序生物有幸参与了此项研究中的ac4C acRIP-seq高通量技术服务。
至此我司已经为合作客户发在Nucleic Acids Research(IF=19.16)、Journal of Hematology & Oncology (IF=23.168)、Advanced Science杂志(IF=17.52)等核心期刊中均提供过ac4C acRIP-seq高通量技术服务以及生信分析。

发表期刊:Nucleic Acids Research
影响因子:19.160
发表时间:2022年8月16日
研究方法:ac4C acRIP-seq、acRIP-qPCR、RIP-qPCR
文章链接:N4-acetylcytidine regulates the replication and pathogenicity of enterovirus 71

01 EV71 RNA的5’ UTR中含有ac4C修饰,且宿主NAT10表达模式发生改变
为了研究EV71 RNA是否含有ac4C修饰,作者大规模纯化EV71 RNA,并通过UPLC-MS/MS进行定量,发现病毒粒子的EV71 RNA中ac4C残基占总胞苷的0.201%。此外,针对EV71基因组RNA进行acRIP-qPCR分析,发现来自病毒感染细胞和病毒粒子的EV71 RNA都被拉下,而在体外转录的病毒基因组没有被拉下。为了绘制EV71基因组中的ac4C修饰谱,作者进行了ac4C acRIP-seq分析。在EV71基因组的311到406核苷酸(nt)区域发现了一个明显的ac4C峰,该峰位于5’ UTR区IRES内的茎环IV(nt 242-445)。EV71不编码任何具有乙酰转移酶活性的蛋白。为了研究EV71 RNA上的ac4C修饰是否被细胞乙酰转移酶NAT10催化,作者进行了RIP-qPCR实验,结果表明NAT10与EV71 RNA相互作用。此外,过表达或敲低NAT10时,病毒RNA上的ac4C的强度相应增加或降低。综上所述,EV71 RNA上含有ac4C修饰,且由宿主乙酰转移酶NAT10介导。EV71在细胞质中进行复制,而NAT10主要分布在细胞核中,而免疫荧光显示,在EV71存在的情况下,NAT10同时定位于细胞核和细胞质,表明EV71感染改变了NAT10的亚细胞定位。此外,在病毒感染过程中检测到较强的NAT10信号,提示EV71感染可能增强了NAT10的表达。
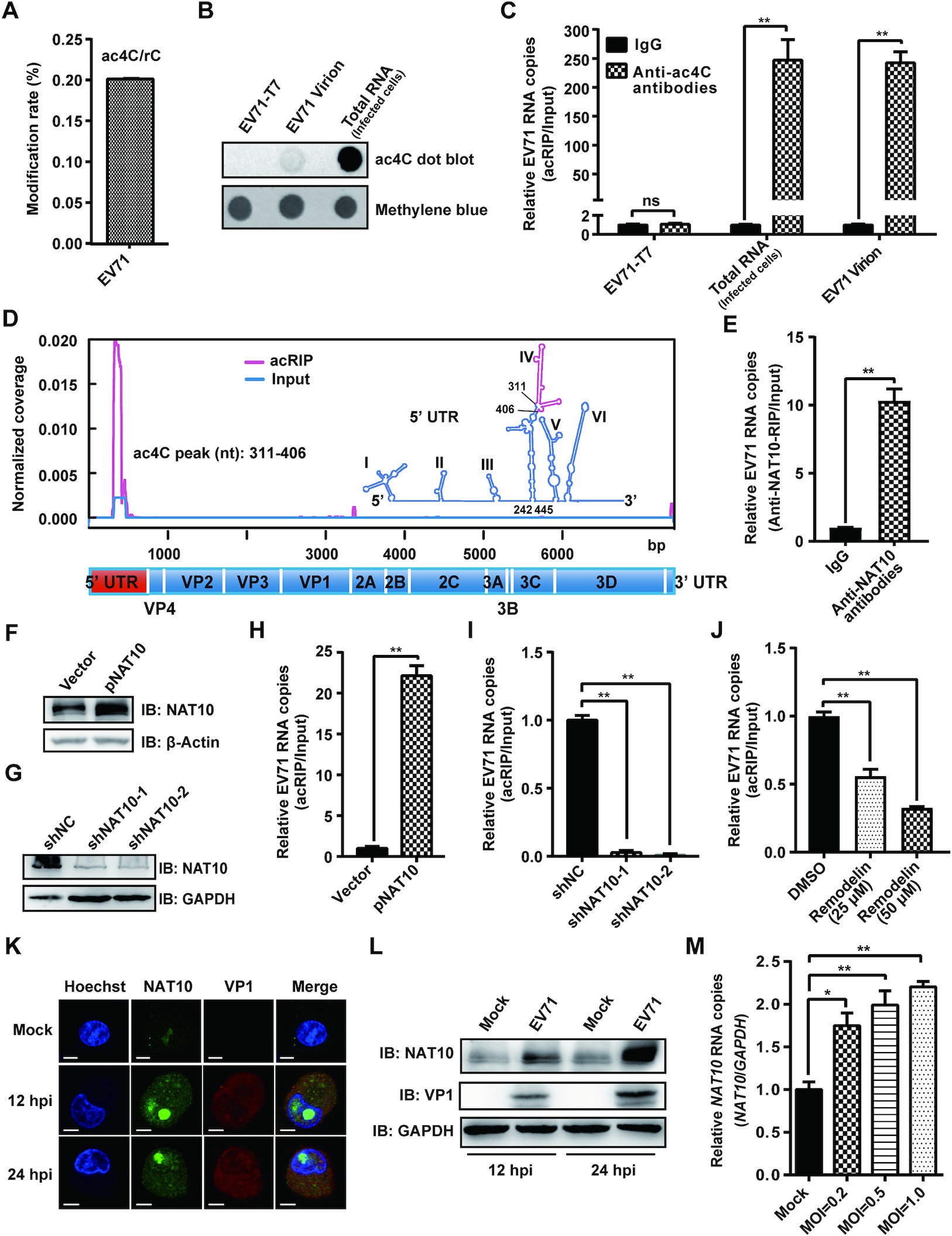
图1. EV71含有ac4C修饰,并改变了NAT10的表达模式
02 5’ UTR区的ac4C修饰促进了EV71的复制
为了研究ac4C是否影响EV71的复制,作者构建了NAT10敲低Vero细胞并感染EV71,发现EV71滴度和RNA拷贝数均显著降低,暗示NAT10促进了病毒的复制。NAT10作用于共识基序5’-CCG-3’,促进中心胞苷的乙酰化。作者为了评估EV71 5’ UTR上的ac4C峰中的两个5’-CCG-3’基序是否被ac4C修饰,构建了EV71 cDNA全长克隆的突变体Mut-331、Mut-350、Mut-331-350以及对照Mut-383。acRIP-qPCR结果显示,与WT EV71相比,ac4C突变病毒粒子和突变病毒感染细胞的EV71 RNA中的ac4C丰度均显著降低,表明这些C残基在EV71基因组中发生乙酰化。且当ac4C位点发生突变时,子代病毒滴度和基因组RNA拷贝数均显著下降,而在shRNA介导的NAT10敲除的细胞中,WT和突变病毒的子代病毒滴度没有差异。这些结果支持了ac4C在EV71复制中起着重要作用。在EV71复制过程中存在正链和负链RNA,与负链RNA相比,下调NAT10更显著地降低了病毒正链RNA的水平,表明NAT10主要影响病毒正链RNA。
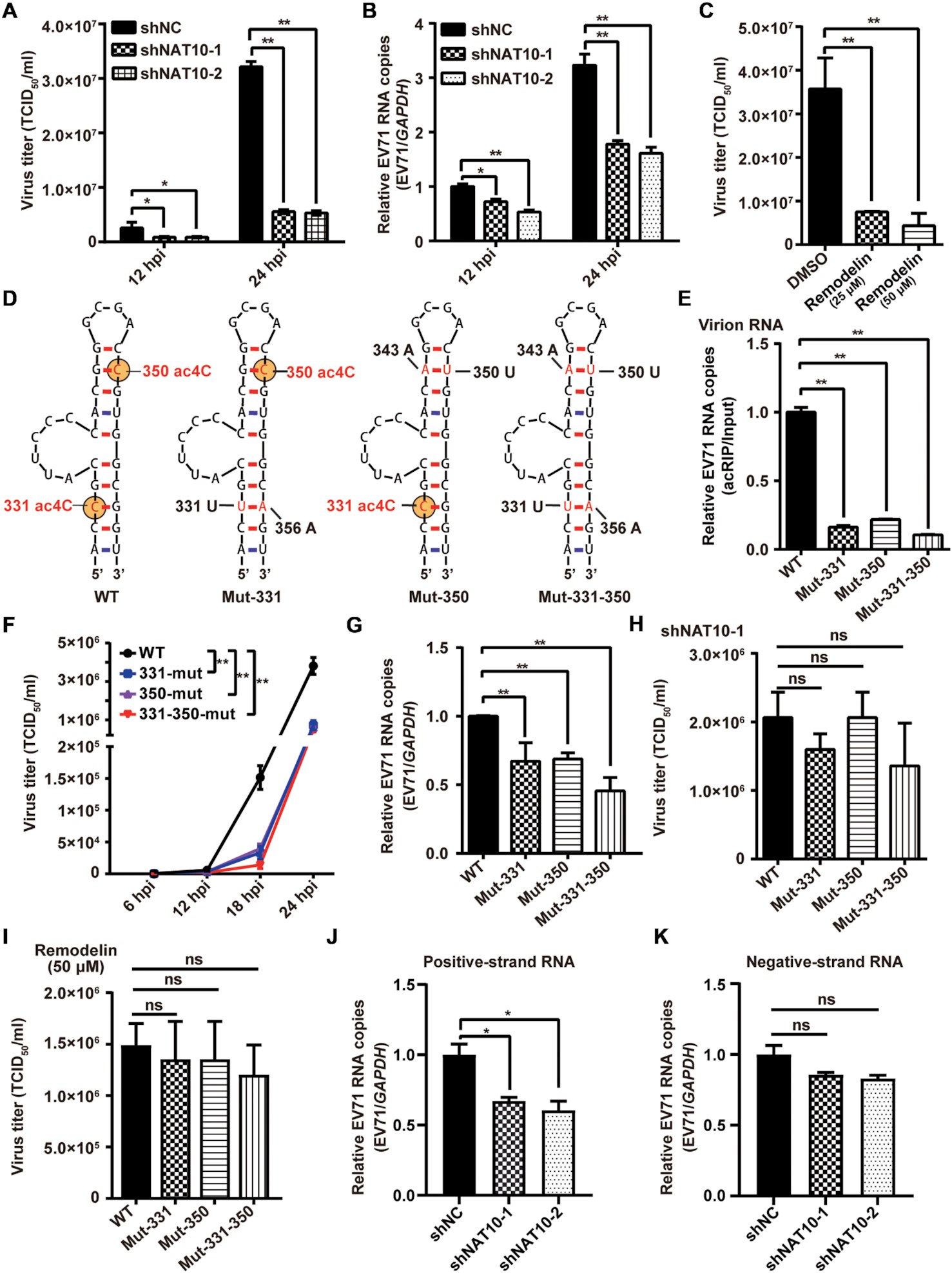
图2. ac4C修饰促进了EV71的复制
03 ac4C修饰通过增强PCBP2与IRES的结合来促进RNA的翻译
EV71 RNA的乙酰化位点位于IRES的茎环IV(nt242-445),对病毒蛋白合成至关重要,因此,ac4C修饰可能影响EV71 RNA的翻译效率。作者用蔗糖密度梯度离心和qRT-PCR检测了在有无NAT10存在的情况下,核糖体对病毒RNA的装载效率。值得注意的是,shRNA介导的NAT10敲除显著抑制了EV71 RNA上的核糖体装载,表明ac4C增强了EV71 RNA的翻译。作者构建了插入EV71 WT或ac4C突变体的5’ UTR区的eGFP报告质粒,并将其转染到RD细胞中,发现EV71 ac4C突变体转染细胞中eGFP的表达减少,进一步表明ac4C提高了EV71 RNA的翻译效率。体外翻译实验同样证实了ac4C促进了EV71 RNA的翻译。接下来,作者通过RIP-qPCR研究了EV71 WT或ac4C突变体IRES与PCBP2和HNRNPK的结合能力,结果表明,ac4C通过选择性地增强PCBP2与IRES的结合来促进病毒的翻译。
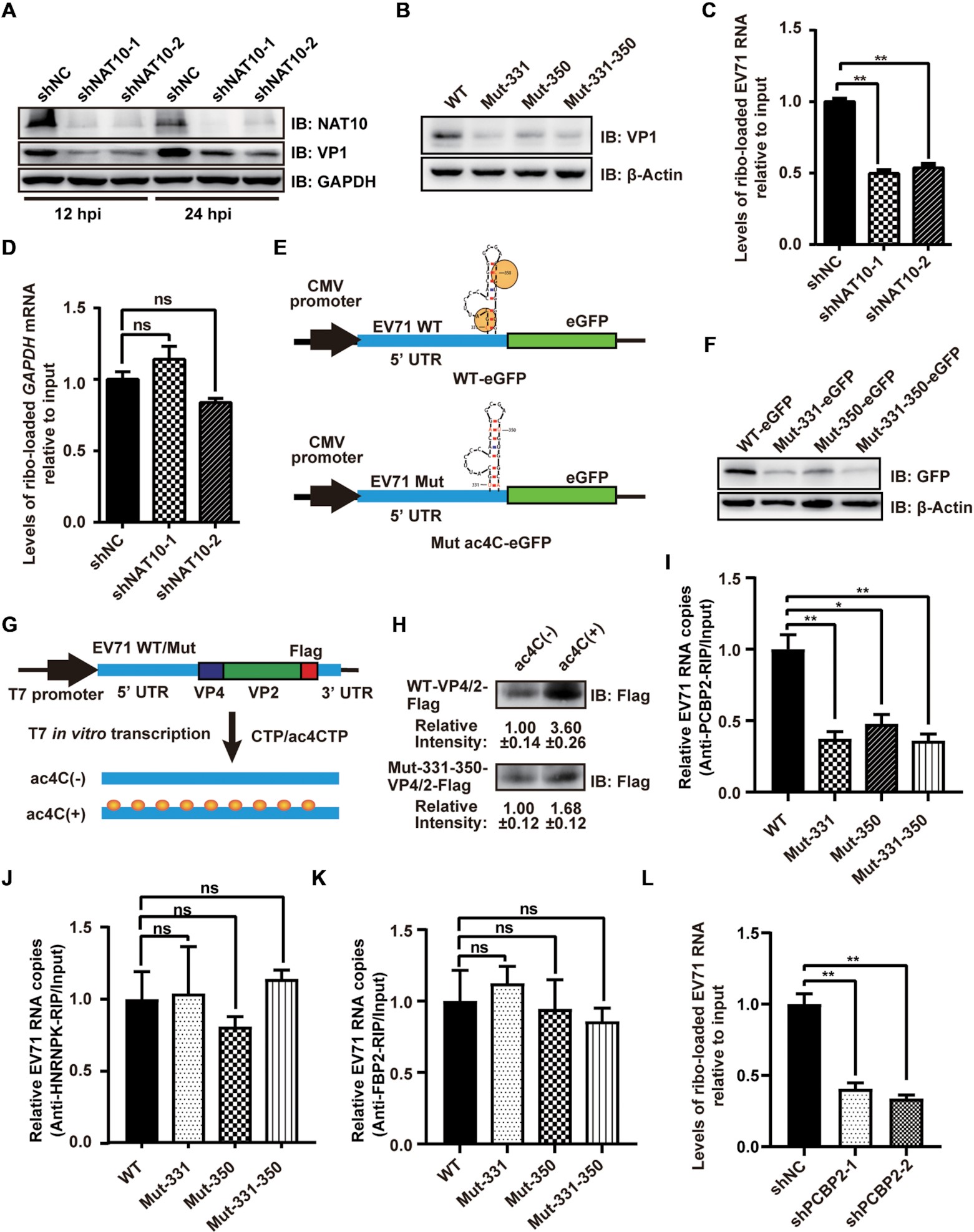
04 ac4C修饰提高了EV71 RNA的稳定性
mRNA的翻译效率和稳定性有着错综复杂的联系,mRNA翻译的减少降低了mRNA的稳定性,反过来降低了翻译效率。为了探讨ac4C修饰是否与EV71 RNA的稳定性有关,作者构建了EV71 WT和ac4C突变体的3D移码突变质粒,并将其转染到Vero细胞中,在不同时间点进行qRT-PCR分析。结果显示,与WT病毒相比,ac4C突变体的RNA降解速度加快。然而,当使用重构蛋白抑制NAT10的功能时,WT病毒和突变病毒之间的RNA降解率没有显著差异。以上结果表明,ac4C修饰影响了RNA的降解效率。值得注意的是,在ac4C修饰的情况下,WT-mut3D和Mut-331-350-mut3D的RNA稳定性都增加了,表明ac4C增强了EV71 RNA的稳定性。接下来,作者探讨了病毒RNA翻译对RNA稳定性的影响。当使用环己酰亚胺处理细胞抑制翻译时,WT-3Dmut RNA的降解加速,而ac4C突变体RNA的降解速率没有明显变化。值得注意的是,PCBP2的敲除导致了病毒RNA的加速降解。这些结果表明,ac4C对EV71 RNA稳定性的影响与病毒RNA的翻译效率有关。
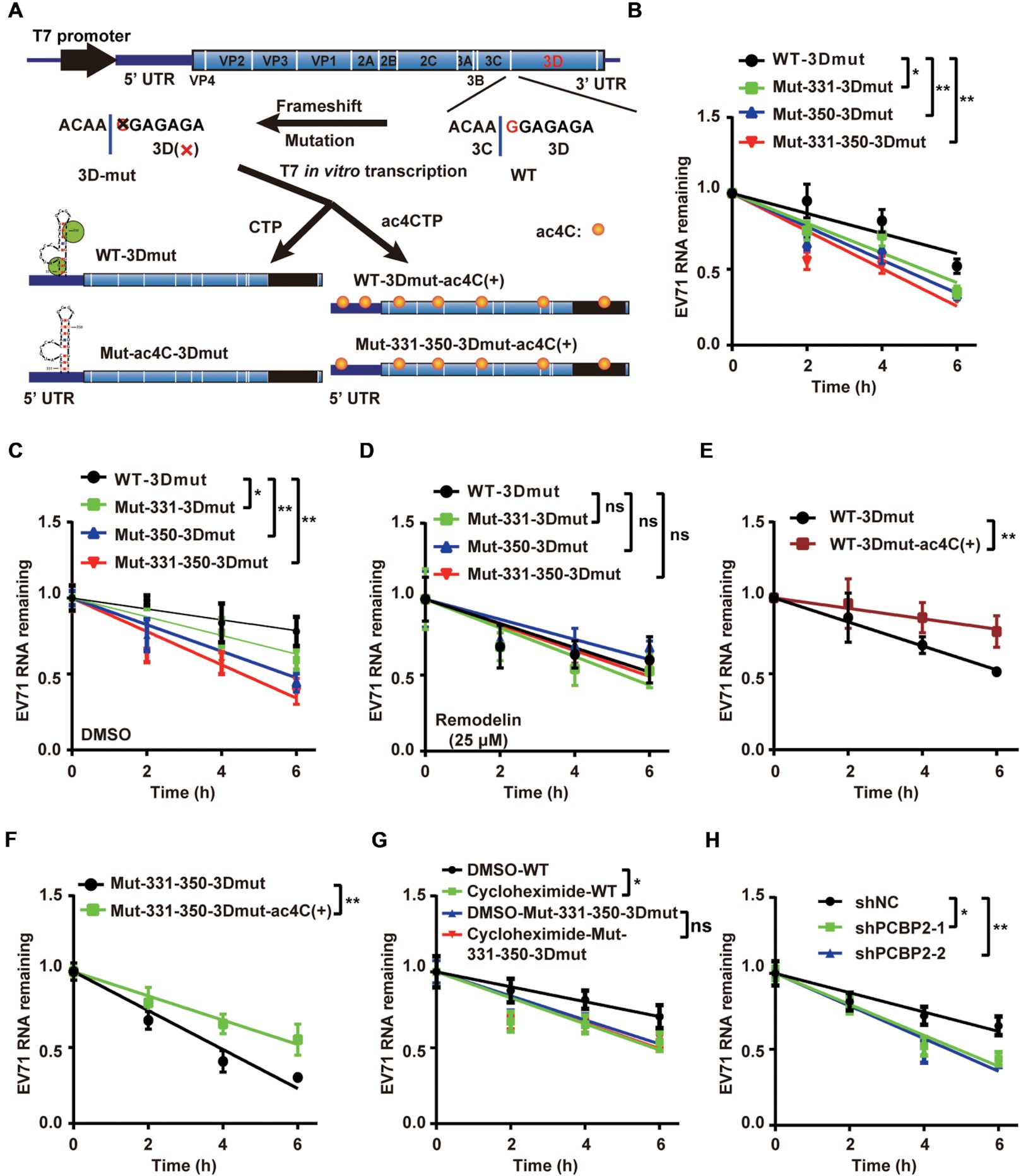
05 ac4C在体内和体外均促进了EV71 RNA与3D的结合
3D是在EV71复制过程中直接结合并合成病毒RNA的关键蛋白。由于乙酰化促进了PCBP2与EV71 RNA的结合,作者接下来评估了ac4C是否影响了3D与病毒RNA的结合。作者通过RIP-qPCR分析与3D结合的RNA,发现3D结合的EV71 RNA通过重构蛋白处理抑制NAT10而减少,表明ac4C修饰影响了病毒RNA与3D的结合。然而,当ac4C突变体病毒与重构蛋白一起培养时,3D结合的RNA并没有明显的变化。这些数据表明,ac4C修饰促进了EV71 RNA与3D的结合。为了探索ac4C修饰是否影响3D与病毒基因组的结合,作者进行了3D体外结合试验实验,结果不仅表明ac4C在体外增强了3D结合,而且表明乙酰化位点的位置对其结合能力具有重要作用。
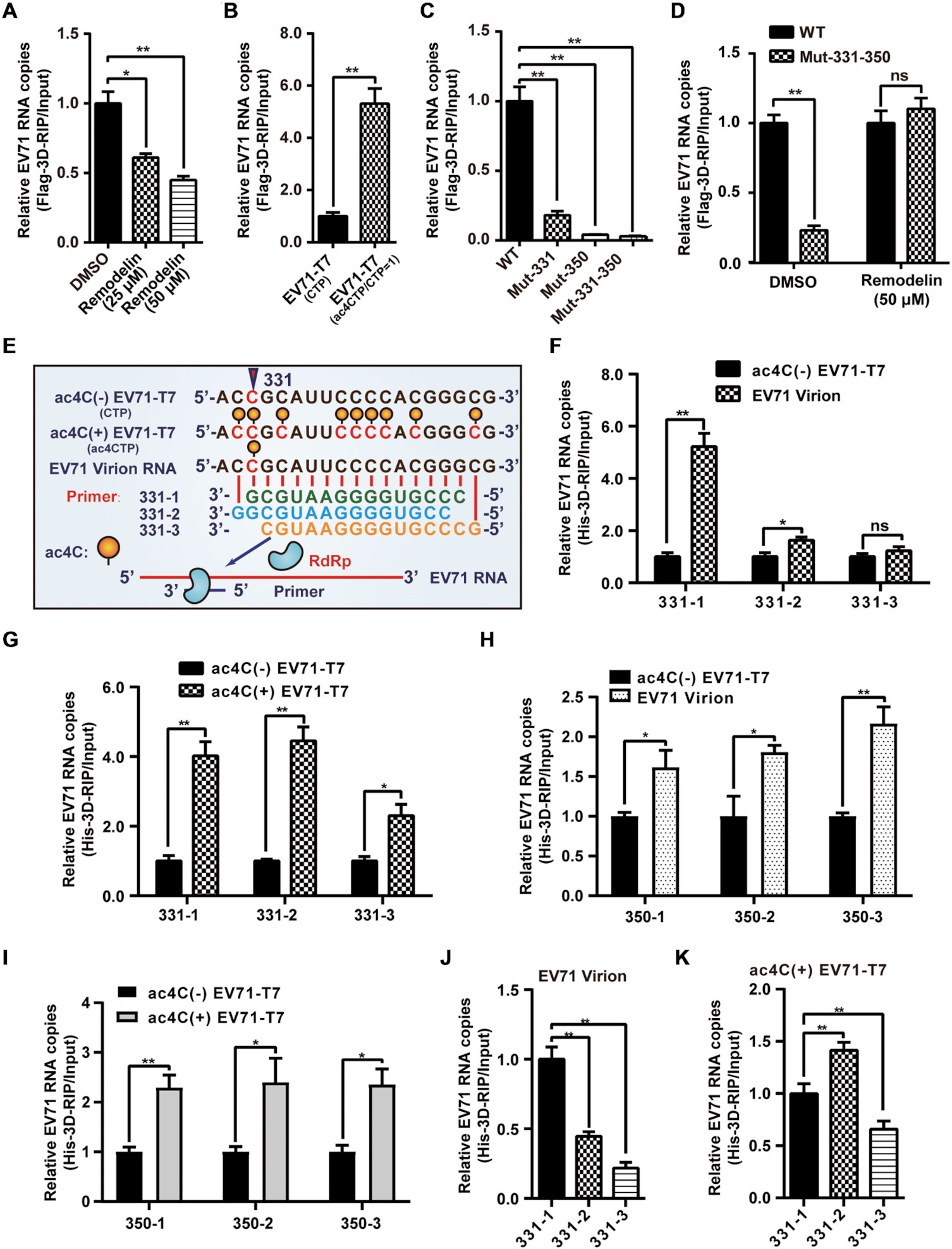
06 EV71 ac4C突变病毒在小鼠体内的致病性降低
为了研究乙酰化是否影响了病毒的致病性,作者用EV71 WT或Mut-331-350感染AG6干扰素受体缺陷小鼠。与感染WT病毒的小鼠相比,感染ac4C突变病毒的小鼠显示出更长的存活时间和更慢的体重减轻,且在感染WT病毒的小鼠中观察到更严重的肢体瘫痪。此外,qRT-PCR分析显示,感染Mut-331-350病毒的小鼠,所有检测器官的肌肉、肠、脑、心、肝、肝、脾、肺和肾中的病毒RNA水平均显著降低。在mut-331-350感染小鼠的肢体肌肉、大脑和肠道中观察到致病性降低,导致肌纤维坏死和神经元损伤减少,肠细胞增生、肠绒毛上皮细胞溶解、绒毛顶端细胞空泡化减少。综上所述,这些结果表明EV71 ac4C突变减少了小鼠的病理损伤。
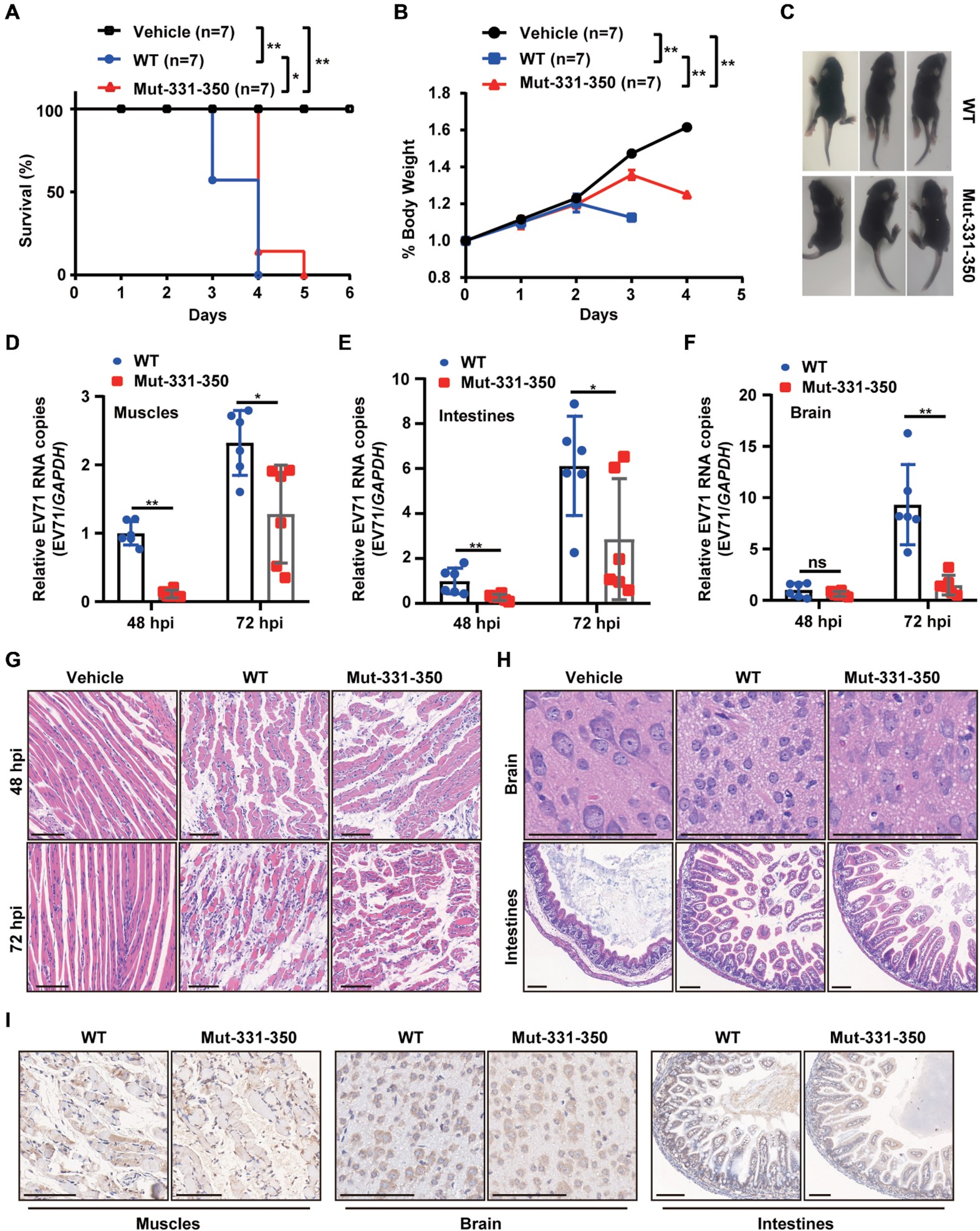
本文运用ac4C acRIP-seq测序技术鉴定到肠道病毒EV71基因组的5’ UTR区IRES内存在ac4C修饰,且由宿主乙酰转移酶NAT10催化产生。该ac4C修饰通过增强PCBP2与IRES的结合来增强病毒RNA的翻译,并提高了RNA的稳定性。此外,抑制NAT10或突变ac4C位点抑制了EV71的复制,且ac4c缺陷突变体EV71在小鼠体内表现出致病性降低。该研究突出说明了ac4C在EV71感染中的重要作用,并为潜在的抗病毒治疗提供了新的方向。
01 ac4C RNA修饰测序
ac4C RNA修饰测序
对ac4C RNA乙酰化,目前最流行的检测手段为acRIP-seq技术,适用于ac4C RNA乙酰化谱研究,快速筛选ac4C RNA乙酰化靶基因。云序可提供mRNA和多种非编码RNA的ac4C测序:
- ac4C 全转录组测序(涵盖mRNA,LncRNA,circRNA)
- ac4C LncRNA测序(涵盖LncRNA和mRNA)
- ac4C Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
- ac4C mRNA测序
- ac4C rRNA测序
- ac4C circRNA测序
- ac4C tRNA测序
02 检测整体ac4C RNA修饰水平
LC-MS/MS检测整体RNA修饰水平
精准高效,可以实现一次检测,9类修饰水平检测,一步到位。
03 ac4C RNA修饰上游酶的筛选
ac4C RNA修饰相关酶PCR芯片
寻找上游直接调控ac4C RNA乙酰化的甲基转移酶。
04 ac4C RNA修饰靶基因验证
acRIP-qPCR
云序提供acRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证靶基因RNA修饰水平。
05 机制互作研究
RIP-seq/qPCR
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
RNA pull down -MS/WB
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
双荧光素酶实验
验证两基因互作,研究相应的分子调控机制。
ChIP-seq
筛选或验证目标蛋白与DNA互作,研究相应的分子调控机制。
优势一:云序累计支持客户发表 84 +篇RNA修饰SCI论文,合计影响因子 814 +,其中RNA乙酰化测序高达3篇论文IF接近20。
优势二:累计完成数千例 RNA甲基化测序样本,全面覆盖医口、农口等各类样本。
优势三:全面检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:独家提供ac4C一站式服务:ac4C整体水平检测、acRIP-seq、acRIP-qPCR验证、RIP和RNA pull down等。
优势五:率先研发超微量acRIP测序技术,RNA量低至500ng起。
优势六:国内最全的RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、m6Am、O8G、ac4C乙酰化和2'-O-甲基化测序。
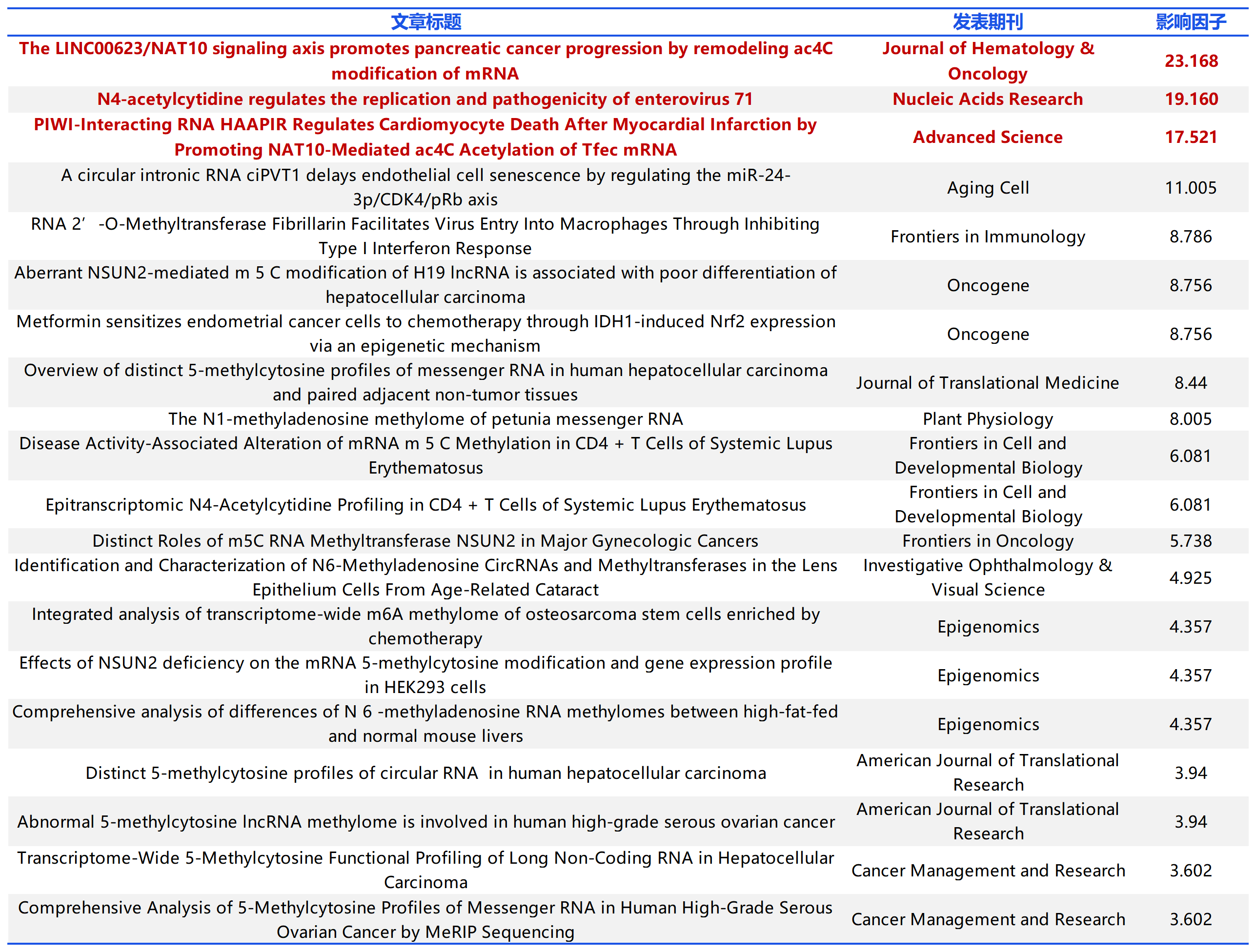
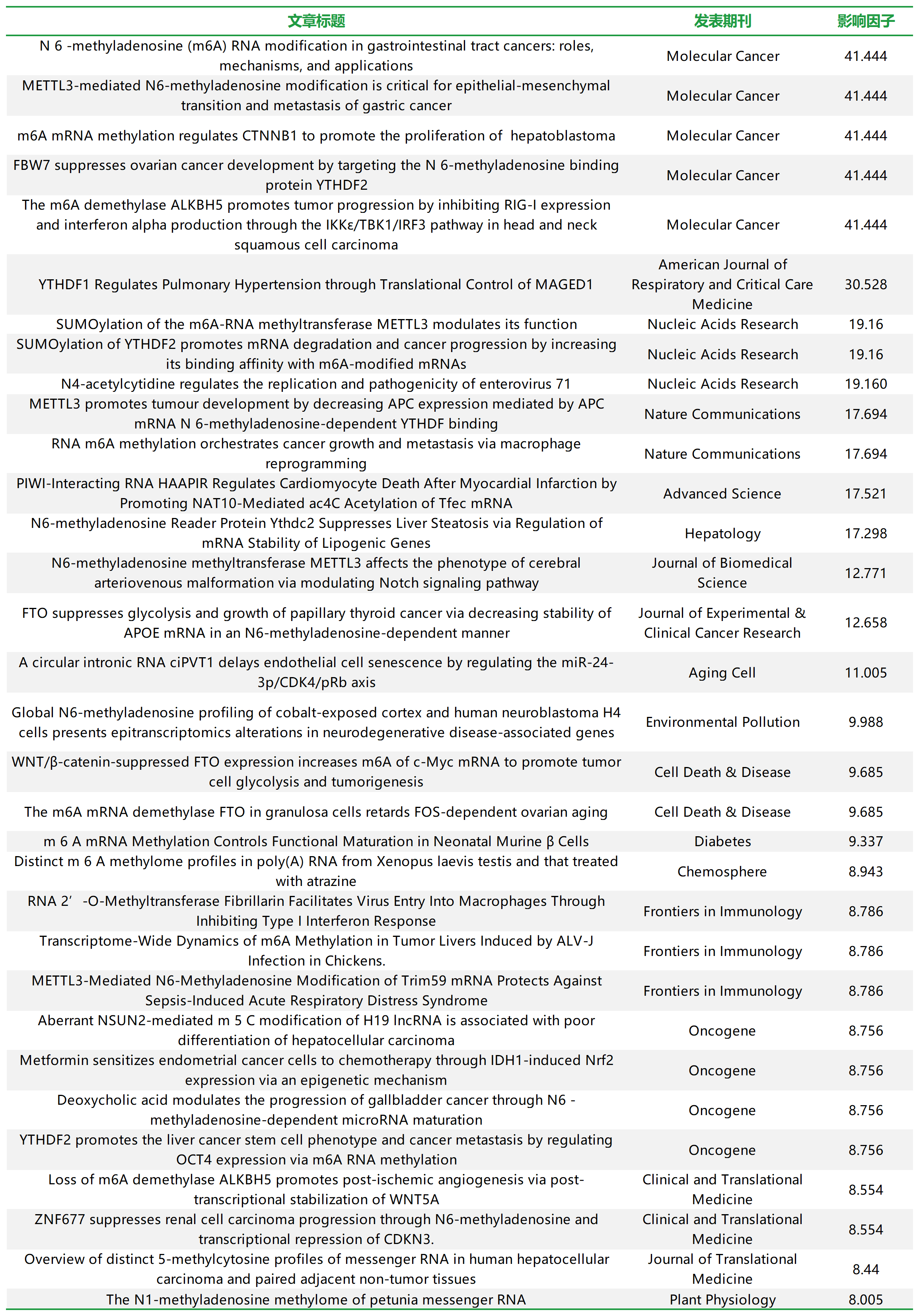
云序客户多种RNA修饰(m6A以外修饰)部分文章列表
相关产品
m5C RNA甲基化测序
m1A RNA甲基化测序
ac4C RNA乙酰化测序
O8G RNA氧化修饰测序
2’-O-RNA甲基化测序
m6Am RNA甲基化测序
LC-MS检测整体ac4C甲基化水平
RNA修饰相关酶PCR芯片
往期回顾
咨询
- 889
- 点赞
- 复制链接
- 举报