
CRISPR/CAS9 sgRNA文库构建和筛选
- MSYL-04
- 铭时医疗
- 浙江省宁波市
- 一个月内
- 按需
- 议价
- 2023-02-20 15:12:45
铭时医疗科技(宁波)有限公司
一键申请试用
咨询
加入意向单
联系方式
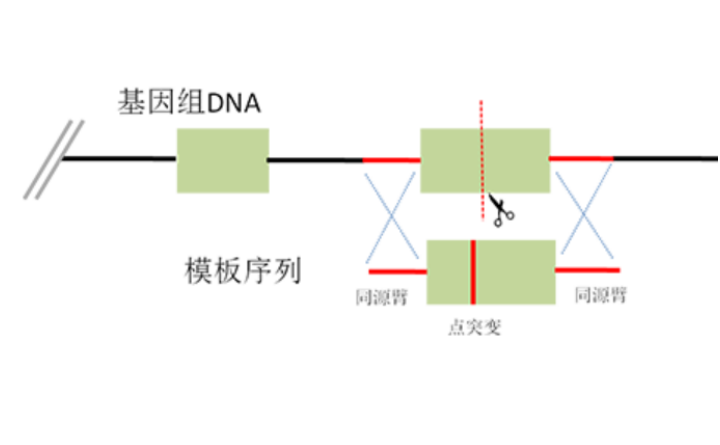
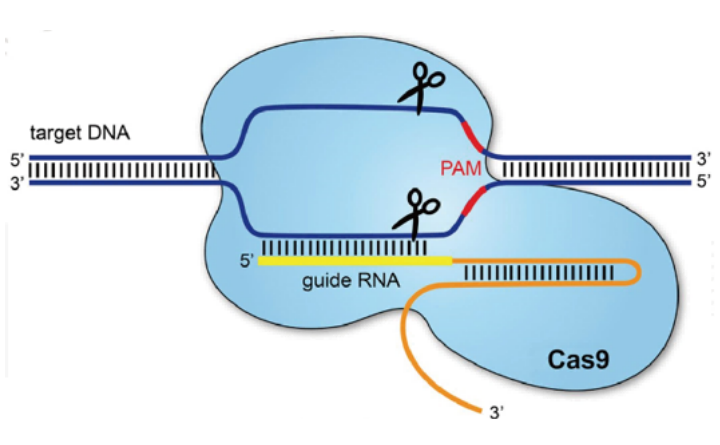
CRISPR/Cas9 基因编辑系统由一个具有DNA双链切割功能的Cas9蛋白 ( 或其他同源蛋白) 和一条具有导向功能的单链RNA (single guide, sgRNA)组成。sgRNA与Cas9 蛋白结合靶向到基因组特定位点,Cas9 切割产生双链断裂(double strand breaks, DSB),DNA序列被切割产生断裂后,引发细胞内部的DNA修复机制,真核细胞同时存在细胞自主性的非同源末端连接(nonhomologous end joining, NHEJ) 或同源重组(homology directed repair, HR)修复,在同源重组修复下,断裂的染色体以另一条完整的姐妹染色单体为模板进行DNA修复,而在非同源重组修复下,断裂出的DNA随机修复,引入新的碱基,使基因产生突变 [1]。已有研究表明,在细胞分裂的各个时期中,细胞自主发生NHEJ 的频率高于HR。
CRISPR/Cas9 基因编辑系统
当sgRNA 靶向单基因时,CRISPR 可视为一种高效的基因编辑工具。但当sgRNA 靶向全基因组序列时,CRISPR 便升级为一种全基因组筛选的工具[2-3]。
利用CRISPR/CAS9技术建立目标物种的全基因组敲除库或者与特定功能相关的基因敲除库,通过功能性筛选和富集以及随后的PCR扩增和深度测序分析,发掘与筛选表型相关的基因,称为CRISPR/CAS9文库筛选。
基于CRISPR/Cas9 筛选的一般流程如下[4]:
CRISPR/CAS9 sgRNA文库构建和筛选可用于:
信号通路相关基因的寻找
药物靶点筛选
药物原发性研究以及基因治疗等
信号通路相关基因的寻找
药物靶点筛选
药物原发性研究以及基因治疗等
我们提供以下服务:
CRISPR/Cas9 sgRNA文库构建和筛选:人/小鼠基因组敲除库
CRISPR/Cas9 sgRNA文库构建和筛选:人/小鼠基因组激活库
CRISPR/Cas9 sgRNA文库构建和筛选:根据客户需求定制基因Panel
CRISPR/Cas9 sgRNA文库构建和筛选:人/小鼠基因组激活库
CRISPR/Cas9 sgRNA文库构建和筛选:根据客户需求定制基因Panel
服务流程
参考文献
1.Hsu PD, Lander ES, Zhang F. “Development and applications of CRISPR-Cas9 for genome engineering.” Cell. 2014;157(6):1262-1278.
2.荆耀彬, 汪伟, 张维绮, 刘光慧. CRISPR/Cas9全基因组筛选在生命科学中的应用. 生命科学, 2018, 30(9): 994-1002.
3.Shalem,Ophir, et al. “Genome-Scale CRISPR-Cas9 Knockout Screening in Human Cells.” Science, vol. 343, no. 6166, 2014, pp. 84–87.
4.Kweon, Jiyeon, and Yongsub Kim. “High-Throughput Genetic Screens Using CRISPR-Cas9 System.” Archives of Pharmacal Research, vol. 41, no. 9, 2018, pp. 875–884.
1.Hsu PD, Lander ES, Zhang F. “Development and applications of CRISPR-Cas9 for genome engineering.” Cell. 2014;157(6):1262-1278.
2.荆耀彬, 汪伟, 张维绮, 刘光慧. CRISPR/Cas9全基因组筛选在生命科学中的应用. 生命科学, 2018, 30(9): 994-1002.
3.Shalem,Ophir, et al. “Genome-Scale CRISPR-Cas9 Knockout Screening in Human Cells.” Science, vol. 343, no. 6166, 2014, pp. 84–87.
4.Kweon, Jiyeon, and Yongsub Kim. “High-Throughput Genetic Screens Using CRISPR-Cas9 System.” Archives of Pharmacal Research, vol. 41, no. 9, 2018, pp. 875–884.